Для осуществления мониторинга за особо опасными инфекциями и проведения своевременных противоэпидемических мероприятий актуальной является разработка новых эффективных диагностических наборов. На сегодняшний день все большее распространение получают методы индикации и идентификации возбудителей различных инфекций с использованием ДНК-чипов. Особенностью данной технологии является возможность проведения одномоментного мультилокусного анализа за сравнительно короткое время при высоких уровнях чувствительности и специфичности. Наравне с полимеразной цепной реакцией (ПЦР) ДНК-чипы могут найти применение для идентификации и генетического типирования возбудителей особо опасных инфекционных заболеваний в профильных лабораториях территориального, регионального и федерального уровней.
Целью настоящей работы являлось конструирование и апробация на панели референтных штаммов микроорганизмов набора реагентов, включающего ДНК-чип, для дифференциального выявления возбудителей особо опасных инфекций: сибирской язвы – Bacillus anthracis, чумы – Yersinia pestis, туляремии – Francisella tularensis, холеры – Vibrio cholerae, легионеллеза – Legionella pneumophila, бруцеллеза – Brucella abortus. Выбор данных микроорганизмов для диагностики с помощью ДНК-чипа обусловлен, прежде всего, высокой вероятностью использования их в качестве средств биотерроризма [1, 2].
Материалы и методы
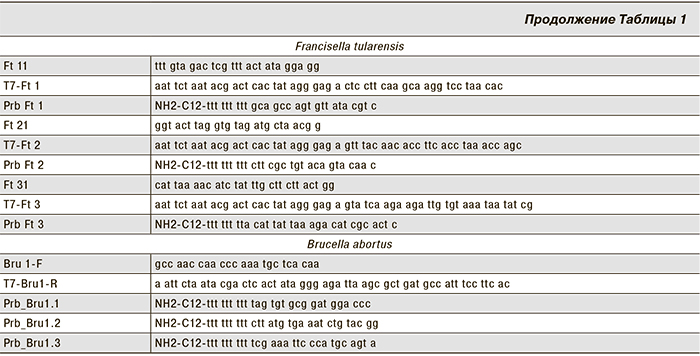
 Выбор структур олигонуклеотидных праймеров и зондов проводили с использованием программного пакета Vector NTI («Invitrogen Corporation», США), а также ресурса, предлагаемого на серверe http://www.basic.northwestern.edu/biotools/oligocalc.html. Все структуры зондов и праймеров сравнивались с помощью программы BLAST (информационный ресурс NCBI) с последовательностями ДНК, депонированными в базе данных GenBank. Олигонуклеотдиные зодны и праймеры были синтезированы в ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора, Москва (далее – ЦНИИЭ). Зонды, предназначенные для последующей иммобилизации на слайдах, содержали аминогруппу на 5’-конце. В состав одного из праймеров каждой пары был включен сайт узнавания для ДНК-зависимой РНК-полимеразы бактериофага Т7. Структуры олигонуклеотидных праймеров и зондов представлены в табл. 1.
Выбор структур олигонуклеотидных праймеров и зондов проводили с использованием программного пакета Vector NTI («Invitrogen Corporation», США), а также ресурса, предлагаемого на серверe http://www.basic.northwestern.edu/biotools/oligocalc.html. Все структуры зондов и праймеров сравнивались с помощью программы BLAST (информационный ресурс NCBI) с последовательностями ДНК, депонированными в базе данных GenBank. Олигонуклеотдиные зодны и праймеры были синтезированы в ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора, Москва (далее – ЦНИИЭ). Зонды, предназначенные для последующей иммобилизации на слайдах, содержали аминогруппу на 5’-конце. В состав одного из праймеров каждой пары был включен сайт узнавания для ДНК-зависимой РНК-полимеразы бактериофага Т7. Структуры олигонуклеотидных праймеров и зондов представлены в табл. 1.
Производство ДНК-чипов
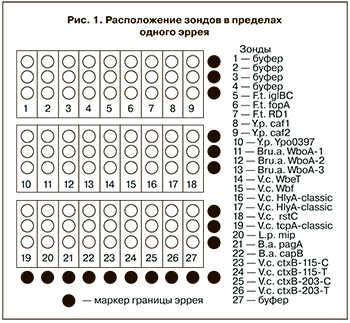
Препараты олигонуклеотидных зондов наносили на микроскопные слайды с поверхностью, модифицированной альдегидными группами, VALS («СЕL Associates Inc.», США) методом бесконтактной печати с помощью пьезоэлектрического робота для микропечати S3 («Scienion AG», Германия) в виде индивидуальных пятен (спотов) диаметром 350 мкм в трех повторах. Олигонуклеотидные зонды и контрольные споты были нанесены в пределах каждой из двенадцати зон (эрреев) на слайде идентичным образом (рис. 1). Далее слайды инкубировали в течение ночи при комнатной температуре, обрабатывали свежеприготовленным 0,25% раствором боргидрида натрия, промывали 0,2% раствором додецилсульфата натрия (SDS), затем дистиллированной водой и высушивали.
Материал для исследования
Для изучения аналитической чувствительности и специфичности ДНК-чипа были использованы штаммы следующих микроорганизмов: Bacillus anthracis (13 штаммов), Yersinia pestis (8), Francisella tularensis (9), Vibrio choleraе (9), Legionella pneumophilla (3), Brucella abortus (1), Bacillus cereus (1), Bacillus subtilis (1), Yersinia psedotuberculosis (1), Yersinia enterocolitica (1), Escherichia coli (2), Vibrio parahaemolyticus (1), Vibrio vulnificus (1), Vibrio mimicus (1). Данные штаммы были предоставлены ФБУН «Государственный научный центр прикладной микробиологии и биотехнологии» Роспотребнадзора (Оболенск) и ФКУЗ Российский научно-исследовательский противочумный институт «Микроб» (Саратов; далее – РосНИПЧИ «Микроб»). Межлабораторные испытания ДНК-чипа проводили в РосНИПЧИ «Микроб» на панелях штаммов, включавших 8 штаммов B. anthracis, по одному штамму B. cereus, B. subtilis и megatherium, 7 штаммов Y. pestis, 1 штамм Y. enterocolitica, 1 штамм Y. psedotuberculosis, 8 штаммов F. tularensis, по одному штамму B. abortus, B. melitensis, B. suis, B. ovis, B. canis и B. neotomae, 10 штаммов V. cholerae, по одному штамму V. parahaemolyticus, V. mimicus и V. vulnificus, 3 штамма L. pneumophila и 1 штамм E. coli. Взвеси бактериальных клеток были приготовлены с использованием отраслевого стандартного образца мутности ОСО 42-28-59-86П ФГБУ «Научный центр экспертизы средств медицинского применения»» Минздрава России. Работы проводили в соответствии с МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I–IV групп патогенности» и СП 1.3.1285-03 «Безопасность работы с микроорганизмами I–II групп патогенности (опасности)».
Процедура проведения анализа
Выделение ДНК из исследуемых материалов проводили с помощью комплекта реагентов «РИБО-преп» производства ЦНИИЭ согласно рекомендациям производителя. Дальнейшие этапы проводили с использованием разработанного набора.
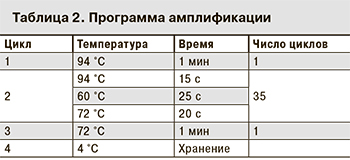
ПЦР-амплификацию проводили в формате «мультипрайм». Праймеры группировали в 2 пробирки по 10 пар праймеров в каждой. Таким образом, для каждого из исследуемых образцов проводилось по 2 реакции амплификации. В состав реакционной смеси входили 10 мкл ПЦР-смеси-2-blue (ЦНИИЭ), 2,5 мкл 10х dNTP-mix (ЦНИИЭ), по 0,08 мкМ прямого и обратного праймеров и бетаин до конечной концентрации 1 М. Объем реакции составлял 25 мкл. Амплификацию проводили в термоциклере Терцик («ДНК-Технология», Россия) по протоколу, представленному в табл. 2.
Транскрипцию ПЦР-продукта проводили с использованием ДНК-зависимой РНК-полимеразы бактериофага Т7. В состав реакционного буфера входили 40 мМ Трис-НСl-буфера (pH = 8,0), 6 мМ хлорида магния, 10 мМ хлорида натрия, 10 мМ DTT, по 0,25 мМ ATP, CTP и GTP (ЗАО «Биосан», Россия), 0,2 мМ UTP (ЗАО «Биосан», Россия), 0,05 мМ biotin-UTP («ДНК-Синтез», Россия), 50 ед. ДНК-зависимой РНК-полимеразы фага T7 (ЦНИИЭ). Для проведения транскрипции в реакцию брали по 10 мкл неочищенного ПЦР-продукта из каждой амплификационной пробирки при общем объеме реакции 50 мкл. Реакцию проводили в течение 40 мин при 37 оС.
При постановке гибридизации для разделения ДНК-чипа на эрреи использовали рамки-трафареты «16 well incubation chamber» («Whatman», Германия), слайды с трафаретами помещали в рамки-держатели «FAST-Frame» («Whatman», Германия). В ячейку вносили 80 мкл предварительно прогретой гибридизационной смеси, состоящей из 10 мкл продукта транскрипции, 30 мкл дистиллированной воды и 40 мкл двукратного гибридизационного буфера. Состав буфера: 600 мМ хлорида натрия, 60 мМ цитрата натрия (4х SSC; pH = 7,0), 20% формамид 40 мМ ЭДТА, 1% SDS. Гибридизацию проводили в течение 40 мин при температуре 37 оС и 300 об/мин с использованием термостатируемого шейкера («Biosan», Латвия), затем содержимое из эрреев убирали с помощью вакуумного медицинского отсасывавателя и отмывали слайды сначала 5х SSC (pH = 7,0) с добавлением 0,1% твина-20, а затем 2х SSC (pH = 7,0) с добавлением 0,1% твина-20. После отмывки слайды инкубировали со стрептавидином, меченым флуоресцирующим красителем Cyanine-5 («Имтек», Россия) в конечной концентрации 2,5 мкг/мл в течение 30 мин при 37 оС и 300 об/мин с использованием термостатируемого шейкера, затем промывали ДНК-чипы раствором PBS-T, далее ополаскивали дистиллированной водой и высушивали.
Учет и интерпретация результатов анализа
После проведения гибридизации слайды сканировали на флюоресцентном лазерном анализаторе для биочипов MArS («Ditabis», Германия) на канале Red (возбуждение при 635 нм, поглощение при 670 нм). Уровни флуоресцентных сигналов от каждого спота оценивали с помощью программы ScanArray Express («Perkin Elmer», США). С помощью специализированного программного обеспечения для интерпретации сигналов флуоресценции «SCAN ARRAY PARSER», разработанного в ЦНИИЭ, вычисляли средние значения флуоресценции, критический уровень флуоресценции (cut off) и коэффициенты позитивности (КП) для каждого исследуемого биомаркера. Если для определенного олигонуклеотидного зонда КП превышал 1,2, результат гибридизации на чипе считали положительным для соответствующего этому зонду фрагмента генома возбудителя.

Подтверждение результатов гибридизации
Специфичность результатов гибридизационного анализа подтверждали с помощью электрофоретической детекции продуктов ПЦР в 1,5% агарозном геле с добавлением бромистого этидия и последующего капиллярного секвенирования фрагментов на приборе Genetic Analyzer ABI Prism 3500xl («Life Technologies», США).
Результаты и обсуждение
В ходе работы в ЦНИИЭ был разработан диагностический набор, включающий ДНК-чип, для одновременного выявления маркеров возбудителей особо опасных инфекций. С целью повышения эффективности гибридизации на ДНК-чипе нами была дополнительно введена стадия транскрипции. На этой стадии с помощью фермента ДНК-зависимой РНК-полимеразы бактериофага Т7 происходит синтез одноцепочечной РНК на матрице двухцепочечных фрагментов ДНК, полученных на этапе ПЦР-амплификации, с одновременным встраиванием рибонуклеозидтрифосфатов, меченых биотином. Такой подход позволяет сократить длительность гибридизации материала на ДНК-чипе и увеличить чувствительность анализа.
Комбинации праймеров для проведения реакций ПЦР-амплификации и зондов для иммобилизации на слайдах были подобраны с учетом особенностей строения геномов выявляемых патогенов.
Известно, что вирулентные штаммы Bacillus anthracis содержат одновременно 2 плазмиды: рХО1 и рХО2 [3, 4], в то время как штаммы, лишенные какой-либо из плазмид, являются авирулентными. Поэтому в качестве основных мишеней для ПЦР нами были выбраны ген capB, находящийся на плазмиде рХО2, и ген pagA (рХО1). Таким образом, при исследовании образцов, подозрительных на инфицированность сибиреязвенным микробом, могут быть получены следующие результаты гибридизации:
- pagA+ capB+ – вирулентные штаммы B. anthracis (рХО1+, рХО2+);
- pagA– capB+ – авирулентные штаммы B. anthracis (рХО1–, рХО2+);
- pagA+ capB– – авирулентные штаммы B. anthracis (рХО1+, рХО2–).
Для определения патогенности и таксономической принадлежности штаммов холерного вибриона, полученных из инактивированных культур, а также из клинического и биологического материала, были выбраны следующие локусы в геноме возбудителя:
- гены ctxA, ctxB, кодирующие синтез соответственно А- и В-субъединиц холерного энтеротоксина и являющиеся маркерами патогенности [5, 6]. Кроме того, анализ структуры гена ctxB позволяет выявить измененные и атипичные варианты штаммов эльтор;
- ген tcpA, кодирующий синтез большой субъединицы токсин-корегулируемых пилей адгезии, считается маркером эпидемической значимости;
- гены wbeT и wbfR, входящие в состав кластера генов, отвечающих за синтез О-антигена групп О1 и О139 соответственно [7, 8];
- ген hlyA, кодирующий термостабильный гемолизин, присутствующий в геноме как вирулентных, так и авирулентных штаммов V. cholerae серогрупп O1 и не-O1, позволяет провести различие между холерными и нехолерными вибрионами, а также между классическими биварами и биоварами эльтор V. cholerae за счет делеции размером 11 п.н. у классических штаммов;
- гены rstC и rstR, регулирующие транскрипцию профага CTXф. Структура локуса rstR существенно отличается у биоваров классического и эльтор. Фрагмент rstC присутствует в составе профага только у штаммов эльтор. Таким образом, оба эти гена позволяют дифференцировать штаммы эльтор и классические между собой.
Для индикации штаммов возбудителей чумы был выбран фрагмент локуса Ypo0397, расположенного на хромосоме и являющегося высокоспецифичным для Yersinia pestis. Существуют капсульные и бескапсульные штаммы Yersinia pestis; бескапсульные характеризуются, как правило, пониженной вирулентностью. Синтез капсулы у возбудителей чумы кодируется плазмидой pFra, поэтому для дифференциации капсульных и бескапсульных штаммов были выбраны фрагменты caf1 и caf1M, расположенные на плазмиде рFra [9].
Для возбудителя туляремии были использованы зонды, гомологичные специфическим участкам гена iglBC (отвечает за синтез белков, необходимых для персистенции возбудителя в макрофагах), гена fopA (кодирует белок наружной мембраны), участок гена RD1 (общий для всех подвидов) [10].
 Для индикации Legionella pneumophila были синтезированы зонды и праймеры, позволяющие определить наличие фрагмента гена mip, который кодирует синтез белка размером 24 кДа, являющегося фактором вирулентности возбудителей [11, 12].
Для индикации Legionella pneumophila были синтезированы зонды и праймеры, позволяющие определить наличие фрагмента гена mip, который кодирует синтез белка размером 24 кДа, являющегося фактором вирулентности возбудителей [11, 12].
С целью определения наличия в исследуемом материале возбудителей бруцеллеза были выбраны и синтезированы зонды и праймеры на фрагмент гена wboA, кодирующего выработку О-антигена и идентичного для штаммов B. melitensis, B. abortus и B. suis [13], наиболее часто вызывающих развитие заболевания у человека.
Разработанный в ЦНИИЭ набор реагентов для индикации возбудителей особо опасных инфекций в формате ДНК-чипа включает все необходимые реагенты для проведения стадий ПЦР-амплификации, транскрипции и гибридизации. Набор предназначен для анализа 48 образцов (включая контрольные). В состав набора входят: ПЦР-смесь 1а, ПЦР-смесь 1b, 5М бетаин и ПЦР-смесь-2-blue для проведения ПЦР-амплификации; 5-кратный транскрипционный буфер, rNTP (5-кратый раствор рибонуклеозидтрифосфатов) и Т7-полимераза для проведения транскрипции; ДНК-чипы, гибридизационный буфер 2х, концентрированный раствор стрептавидина, конъюгированного с Cyanine-5, раствор для разведения стрептавидина и концентрированные отмывочные растворы для проведения стадии гибридизации, а также инструкция к набору. Общее время анализа, без учета стадии выделения ДНК из исследуемого материала, составляет 5,5 ч.
С целью апробации диагностического набора в ЦНИИЭ были изготовлены 3 экспериментально-производственные серии. Межлабораторные испытания ДНК-чипа для выявления возбудителей особо опасных инфекций проводились на базе РосНИПЧИ «Микроб». В ходе межлабораторных испытаний, проводимых на очищенных инактивированных культурах возбудителей, неспецифической амплификации и гибридизации не наблюдалось. Аналитическая чувствительность набора реагентов в отношении выявления Bacillus anthracis по результатам проведенных испытаний составляла от 1×103 м.к./мл в образце, при этом ДНК-чип позволял дифференцировать моноплазмидные и биплазмидные штаммы B. anthracis (рис. 2 А, см. на вклейке). В отношении возбудителей холеры набор позволял выявлять присутствие Vibrio cholerae в концентрации от 1×103 м.к./мл, а также характеризовать обнаруженные штаммы с точки зрения их патогенности, принадлежности к серогруппам О1, О139 или не-О1 не-О139, принадлежности к классическому, эльтор или атипичному биовару эльтор (рис. 2 Д, см. на вклейке). Аналитическая чувствительность в отношении возбудителей чумы и туляремии составляла от 1×103 м.к./мл в образце, при этом набор позволял дифференцировать капсульные и бескапсульные штаммы Yersinia pestis (рис. 2Б, см. на вклейке). Аналитическая чувствительность в отношении выявления возбудителей легионеллеза и бруцеллеза по результатам испытаний составила от 1×104 м.к./мл в образце.
Таким образом, разработанный набор реагентов, основанный на проведении мультипраймерной ПЦР с последующей транскрипцией и гибридизацией материала на ДНК-чипе, позволяет не только выявить в исследуемом клиническом или биологическом материале за одну постановку сразу несколько возбудителей, но и проводить их генетическую характеристику. Показатели чувствительности и специфичности набора реагентов, продемонстрированные в результате межлабораторных испытаний на панели гомологичных и гетерологичных штаммов микроорганизмов, свидетельствуют о возможности использования данного способа диагностики в отношении возбудителей особо опасных инфекционных заболеваний в профильных лабораториях разных уровней. В дальнейшем спектр выявляемых патогенов или их генетических маркеров предполагается расширить за счет размещения в пределах одного эррея бóльшего количества зондов.
* * *
Работа выполнена в рамках федеральной целевой программы «Национальная система химической и биологической безопасности Российской Федерации (2009–2014 годы)».


